Gene in Mitochondrien stillgelegt
Die Genexpression in Mitochondrien zu beeinflussen, war bisher nicht möglich. Forscher aus Göttingen haben nun einen innovativen Ansatz entwickelt, der erstmals gezielte Einblicke in mitochondriale Erkrankungen erlaubt.
Mitochondrien besitzen ihr eigenes Genom – dreizehn zentrale Proteine der Atmungskette werden von der mtDNA kodiert. Bisher war es technisch nicht möglich, gezielt in die Expression dieser Proteine einzugreifen, ohne das Genom zu verändern. Einem Forschungsteam um Prof. Dr. Peter Rehling, Direktor des Instituts für Zellbiochemie der Universitätsmedizin Göttingen (UMG), ist es nun gelungen, die Proteinproduktion in den Mitochondrien lebender Zellen mit Hilfe molekularer Chimären gezielt zu unterbinden. Die in Science vorgestellte neue Technologie ermöglicht fundamentale Einblicke in die zelluläre Energieproduktion und bietet neue Ansätze zur Erforschung von Krankheitsmechanismen.
Funktionsstörungen der Atmungskette und der beteiligten Proteine führen zu mitochondrialen Erkrankungen wie Kardiomyopathien, progressiver Muskelschwäche oder dem Leigh-Syndrom, einer schweren neurodegenerativen Erkrankung. Umso wichtiger ist es, einen Ansatzpunkt für neue Therapien zu finden. Die Göttinger Gruppe untersucht im ERC-Projekt MiXpress die mitochondriale Genexpression. „Um die molekularen Mechanismen der Proteinproduktion in Mitochondrien zu verstehen, brauchen wir experimentelle Ansätze, die es uns erlauben, die einzelnen Schritte zu beeinflussen”, so Rehling. Die CRISPR-Genschere kommt dafür nicht in Betracht, weil die Mitochondrienmembran unüberwindbar ist.
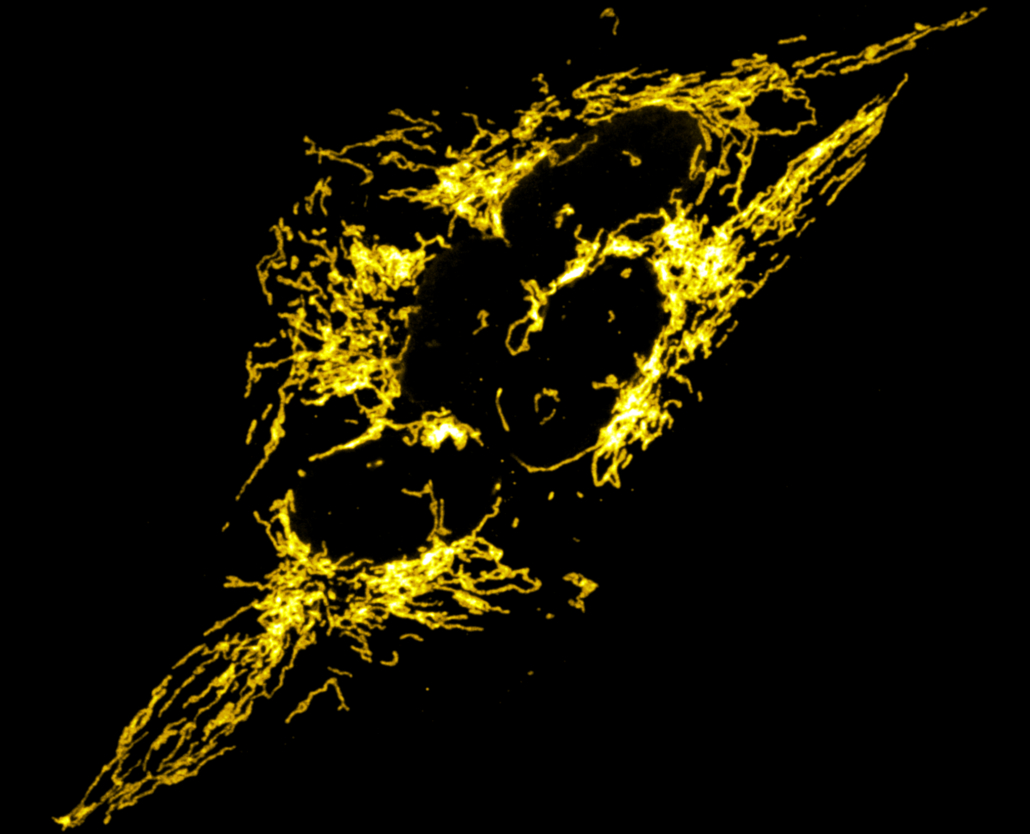
In der aktuellen Studie wurden stattdessen Morpholino-Chimären eingesetzt, die dank ihrer mitochondrialen Zielsequenz (MTS) in die Organellen gelangen können. Der Ansatz wirkt einfach, war aber methodisch lange nicht umsetzbar. Morpholino-Oligomere sind stabile Nukleinsäure-Analoga, die Ziel-RNAs durch spezifische Bindung blockieren. Über eine Click-Reaktion wurden die chemisch modifizierten Oligomere mit der MTS verknüpft und durch Lipofektion in Herzmuskel- und Leberzellen eingeschleust. Um in den Mitochondrien nachverfolgt werden zu können, wurden sie darüber hinaus mit FITC fluoreszenzmarkiert. Erstmals konnte so die Expression mitochondrialer Gene erfolgreich ausgeschaltet werden – im Multiplexing sogar für mehrere Ziel-mRNAs gleichzeitig. So ließ sich auch die Genregulation über die Untereinheiten aller fünf Komplexe der Atmungskette hinweg systematisch analysieren. In Zusammenarbeit mit dem Göttinger Exzellenzcluster Multiscale Bioimaging (MBExC) konnte die Stilllegung durch quantitative NanoString-Genexpressionsanalyse sowie moderne Imaging-, Massenspektrometrie- und Proteomik-Methoden nachgewiesen werden.
Überraschenderweise zeigten sich trotz Blockade zentraler mitochondrialer Untereinheiten keine unmittelbaren Kaskadeneffekte innerhalb der Atmungskette. Statt eines Systemzusammenbruchs offenbarte sich ein fein abgestimmtes Kompensationsnetzwerk mit komplexen regulatorischen Anpassungen – sowohl im mitochondrialen als auch im nukleären Genom. Dabei wurden auch neue Regulatorproteine identifiziert, die an der Koordination zwischen beiden Genomen beteiligt sind.
Die neue Technologie eröffnet somit völlig neue Möglichkeiten, Einblicke in die Entstehung mitochondrialer Erkrankungen zu gewinnen – und darauf aufbauend neue Therapien zu entwickeln. Eine organellspezifische RNA-Blockade könnte künftig gezielt dort ansetzen, wo mitochondriale Erkrankungen ihren Ursprung haben.

 CSEM
CSEM Merck KGaA
Merck KGaA Illustration NIH.gov
Illustration NIH.gov